การตรวจทางพันธุกรรมสำหรับทารกระหว่างการตั้งครรภ์ (Prenatal Genetic Test )
พญ. พันธุ์ชิตา พงศ์ศุภสมิทธิ์
รศ.ดร.นพ.วีรวิทย์ ปิยะมงคล
ผู้หญิงตั้งครรภ์ทุกคนควรได้รับทางเลือกในการตรวจสารทางพันธุกรรมของทารกเพื่อตรวจคัดกรองภาวะโครโมโซมมากกว่าหรือน้อยกว่าปกติ (Aneuploidy) และการตรวจเพื่อวินิจฉัยโรคทางพันธุกรรมก่อนคลอด (1) โดยการตรวจทางสารพันธุกรรมนั้นจะแบ่งเป็น การตรวจเพื่อคัดกรอง (Prenatal genetic screening) และ การตรวจเพื่อวินิจฉัย (Prenatal genetic diagnosis) ในปัจจุบันวิธีการตรวจเพื่อคัดกรองได้แก่ การตรวจ Cell free fetal DNA ซึ่งในเป็นวิธีที่นิยมใช้เนื่องจากมีความน่าเชื่อถือมาก และลดการทำหัตถการรุกล้ำแก่ทารกในครรภ์ ในส่วนการตรวจสารทางพันธุกรรมเพื่อการวินิจฉัยที่นิยมใช้ได้แก่ การตรวจ Cytogenetic analysis หรือ karyotyping (Classical cytogenetics/chromosome banding technique), การตรวจ Fluorescence In Situ Hybridization (Molecular cytogenetic), การตรวจ Quantitative fluorescence polymerase chain reaction (QF-PCR), การตรวจ Chromosomal microarray analysis และ การตรวจ Whole genome sequencing หรือ whole exome sequencing โดยการตรวจวิธีนี้จะถูกเลือกทำในบางกรณีเท่านั้น และ ไม่แนะนำให้ใช้เป็นวิธีการตรวจเป็นประจำ สิ่งส่งตรวจนั้นสามารถนำมาจากน้ำคร่ำหรือชิ้นเนื้อรก และทำการเพิ่มจำนวนสารทางพันธุกรรม(DNA) จากสิ่งส่งตรวจโดยใช้วิธีการ Polymerase chain reaction (PCR) (2)
สำหรับการเลือกใช้เทคนิคการตรวจนั้นขึ้นอยู่กับว่าเราต้องการตรวจหาอะไร ซึ่งหลัก ๆ แล้วจะสามารถแบ่งการทดสอบเป็นสามอย่าง คือการตรวจหายีนส์กลายพันธุ์ (mutation detection), การตรวจหาโครโมโซมที่ผิดปกติ cytogenetic studies และ การตรวจ Genotyping method ซึ่งในที่นี่จะขอกล่าวถึงการตรวจที่มีการนำมาใช้กับการตรวจทางพันธุกรรมในทารกระหว่างตั้งครรภ์ (3)
เทคนิคการตรวจทางพันธุศาสตร์
การตรวจ Standard cytogenetic analysis หรือ Karyotype analysis
เป็นการตรวจที่ใช้บ่อยในการหาโครโมโซมที่ผิดปกติ โดยความผิดปกติ หรือการเปลี่ยนแปลงของโครโมโซมที่สามารถตรวจได้จากวิธีนี้มักจะต้องมีขนาดใหญ่ เช่น Translocation, Large deletion หรือ Aneuploidy โดยการทำ Karyotyping นั้นจะสามารถตรวจหาจำนวนโครโมโซมที่ผิดปกติ เช่น กลุ่มโรค Aneuploidy และสามารถตรวจหา Balanced หรือ Unbalanced structural rearrangement ของโครโมโซมที่มีขนาดอย่างน้อย 5-10 Mega base ขึ้นไป(1, 2) โดยความน่าเชื่อถืออยู่ที่ร้อยละ 99 โดยการตรวจควรจะใช้เนื้อเยื่อใดที่มีเซลล์ที่สามารถแบ่งตัวได้ หรือเซลล์ที่สามารถถูกกระตุ้นให้แบ่งเซลล์ได้ (2)
เซลล์ที่สามารถแบ่งตัวได้จะถูกสารเคมีทำให้หยุดค้างอยู่ที่ระยะ metaphase ซึ่งเป็นระยะที่โครโมโซมถูกทำให้หดตัวและหนาแน่น และสามารถมองเห็นลักษณะแถบของโครโมนโซมได้ชัดเจนที่สุด ซึ่งแต่ละโครโมโซมจะมีลักษณะแถบที่เป็นเอกลักษณ์จำเพาะของตัวเองทำให้สามารถแบ่งแยกโครโมโซมแต่ละคู่ได้ และ โครโมโซมที่ถูกย้อมจะเห็นเป็นลักษณะแถบสีสว่างและแถบสีเข้ม โดยเทคนิคการย้อมที่ใช้จะเป็นการย้อมโดย Giemsa staining ซึ่งจะทำให้เห็นเป็นลักษณะ G band และสามารถตรวจหาส่วนที่ขาดหายไป ส่วนที่เกิน หรือ ส่วนที่มีการสลับเรียงตัวใหม่ได้ ความน่าเชื่อของการตรวจ Cytogenetic analysis จะเพิ่มตามจำนวนแถบสีเข้มสว่าง (Bands) ที่ย้อมติด แถบสีเข้มสว่างที่มีความละเอียดสูงในช่วงระยะเมตาเฟส (High resolution metaphase banding) โดยปกติจะมีแถบให้เห็นได้ประมาณ 450-550 แถบต่อโครโมโซมชุดเดี่ยว (Haploid) 1 ชุด (2)
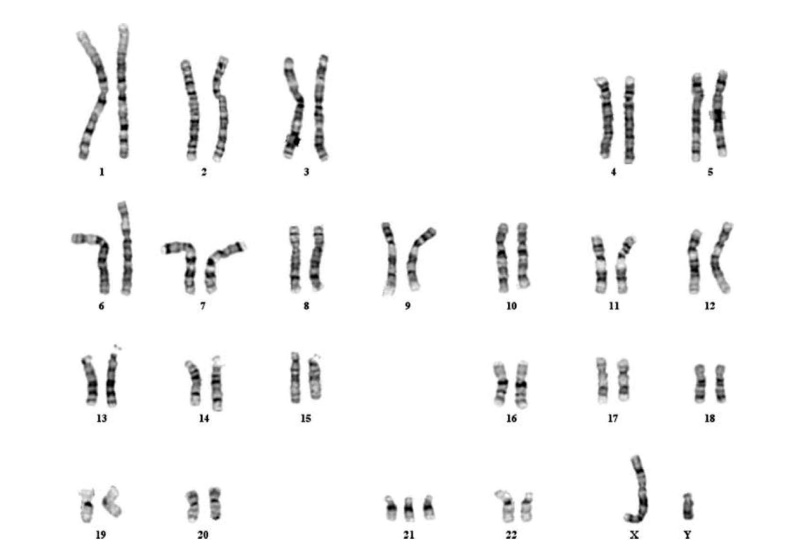
รูปที่ 1 แสดงการตรวจ Standard Karyotype ที่พบว่ามีโครโมโซมคู่ที่ 21 เกินมา 1 แท่ง ในภาวะ Trisomy 21(2)
เนื่องจากเซลล์ที่สามารถแบ่งตัวได้เท่านั้นที่จะสามารถนำมาทดสอบได้ (Metaphase analysis of cultured cell)ดังนั้นความเร็วของผลตรวจจะขึ้นกับความเร็วในการเติบโตของเซลล์ที่นำเพาะเลี้ยง ในน้ำคร่ำจะประกอบไปด้วยเซลล์เยื่อบุ เซลล์เยื่อบุทางเดินอาหาร และ Amniocyte โดยผลตรวจจากเซลล์ดังกล่าวจะได้ภายใน 7-10 วัน ส่วนเซลล์เม็ดเลือดทารก อาจะให้ผลเร็วถึง 36-48 ชั่วโมงแต่ไม่ค่อยนำมาใช้ในทางคลินิก ส่วนในกรณีทารกหลังเสียชีวิต จะทำการตรวจจาก skin fibroblast ซึ่งอาจใช้เวลาถึง 2-3 สัปดาห์เนื่องจากการกระตุ้นให้เซลล์แบ่งตัวนั้นทำได้ยาก(2)
การตรวจ Fluorescence In Situ Hybridization (FISH)
เทคนิคนี้เป็นวิธีที่เร็วในการใช้เพื่อระบุความผิดปกติของโครโมโซมที่มีลักษณะจำเพาะ (Specific chromosome abnormality) และเพื่อยืนยัน Microdeletion หรือ Duplication ที่สงสัย เช่น ภาวะ 22q11.2 microdeletion (DiGorge syndrome) เนื่องจากการตรวจจะใช้เวลาในการทดสอบ 1-2 วัน จึงมักถูกเลือกใช้ในกรณีที่ผลการทดสอบจะเปลี่ยนแปลงแนวทางการรักษาในการตั้งครรภ์ (1, 2)
ในการตรวจ Fluorescence In Situ Hybridization หรือ FISH เซลล์จะถูกตรึงบนสไลด์แก้ว และ DNA probe ที่ถูกติดฉลากสีเรืองแสง (Fluorescent labeled DNA probe) จะไปจับกับโครโมโซมที่ถูก fixed ไว้ โดยที่แต่ละ DNA probe จะมีลำดับ DNA ที่เป็นคู่สมกับลำดับ DNA ของยีนที่สนใจบนโครโมนโซมที่ถูกนำมาทดสอบ หากบนโครโมโซมนั้นมีลำดับ DNAของยีนที่สนใจ probe ก็จะสามารถเข้าจับได้ และสามารถเห็นสีเรืองแสงได้จากกล้อง microscopy ซึ่งจำนวนของสีที่เห็นยังสามารถใช้ประโยชน์ในการเป็นตัวบอกจำนวนโครโมโซม หรือ จำนวนยีน ในเซลล์หนึ่งเซลล์ที่เอามาทดสอบ ว่ามีเกิน หรือ ขาด หรือไม่ โดยที่การตรวจนี้จะไม่ได้ให้ข้อมูลของยีนทั้งโครโมโซมแต่บอกแค่ข้อมูลของส่วนของโครโมโซม หรือยีนที่สนใจเท่านั้น (1-3)
บทบาทในการนำมาใช้ด้าน Prenatal genetic test ของ FISH นั้นคือ การตรวจโครโมโซมในระยะอินเตอร์เฟส(Interphase) ซึ่งเป็นระยะที่มีการสร้างส่วนประกอบต่าง ๆ ของเซลล์เพื่อเตรียมพร้อมสำหรับการแบ่งเซลล์ โดยใช้ลำดับดีเอ็นเอ (DNA sequenced) ที่จำเพาะกับโครโมโซมคู่ที่ 21 , 18, 13, X และ Y เพื่อตรวจจำนวนโครโมโซมเหล่านี้ว่ามีขาด หรือเกินหรือไม่ (2)
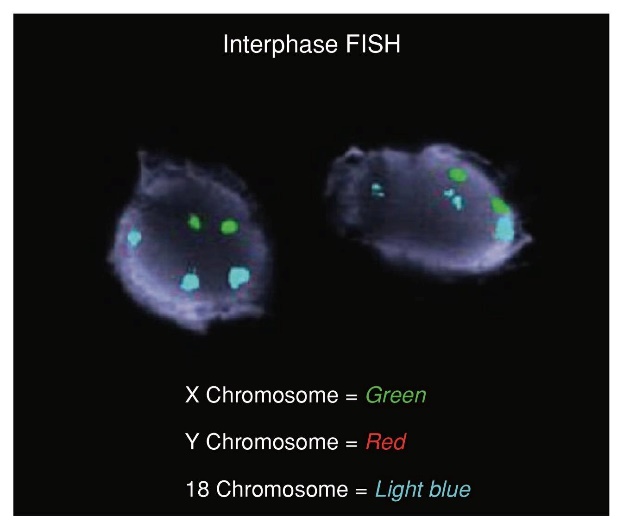
รูปที่ 2 ภาพแสดง Interphase Fluorescence In Situ Hybridization (FISH) โดยการใช้ alpha satellite probe สำหรับ โครโมโซมคู่ที่ 18 (สีฟ้า) , X(สีเขียว) และ Y (สีแดง) ซึ่งในเคสนี้พบสีเขียวสองจุด และสีฟ้าสามจุดในหนึ่งเซลล์ ซึ่งหมายถึงการมีโครโมโซมคู่ที่ 18 เกิน และเป็นทารกเพศหญิง (2)
จากการทดสอบมากกว่า 45,000 ตัวอย่าง พบว่าผลของ FISH และ Standard cytogenetic karyotyping ตรงกันถึง ร้อยละ 99.8 (2) ACOG 2016 ไม่แนะนำให้ใช้ FISH เพียงอย่างเดียวในการวินิจฉัยหากพบผลตรวจผิดปกติ ควรใช้ข้อมูลจาก FISH ร่วมกับการตรวจอย่างน้อยข้อหนึ่งดังต่อไปนี้ การตรวจวินิจฉัยทางพันธุกรรม Karyotyping หรือ การตรวจ Chromosomal microarray หรือ ข้อมูลทางคลินิกที่ช่วยส่งเสริม เช่น การตรวจคัดกรองภาวะ Aneuploidy ที่ผิดปกติ หรือ ภาพถ่ายอัลตราซาวด์ (1)
การตรวจ Quantitative fluorescence polymerase chain reaction (QF-PCR)
เป็นการทดสอบ Polymerase chain reaction ที่มีการสร้าง Amplicon หรือชิ้นส่วน DNA ที่มีลำดับทางพันธุกรรมแบบทำซ้ำโดยตรง (หัวต่อหาง) หรือการทำซ้ำแบบกลับหัว (แบบตัวต่อตัวหรือแบบหางถึงหาง) ซึ่งชิ้นส่วนนี้จะถูกติดฉลากเรืองเเสงไว้ และมีส่วนที่หางที่เป็นจุดสิ้นสุดของการสร้าง DNA และสามารถตรวจหาชิ้นส่วนเหล่านี้โดยใช้ Capillary electrophoresis เทคนิคที่มีการใช้อย่างมากในการทำ QF-PCR คือ การเพิ่มจำนวน Short tandem repeats (STRs) หรือส่วนของ DNA ที่มีลำดับนิวคลีโอไทด์ที่สั้นและที่มีความจำเพาะต่อโครโมโซมซึ่งลำดับนี้จะเป็นชุดซ้ำๆกัน และโดยปกติจะประกอบไปด้วย 2-5 คู่เบสต่อชุด (4)
QF-PCF จะทำโดยการสกัด DNA ออกมาจากน้ำคร่ำ หรือจากชิ้นเนื้อรก โดย DNA จากสิ่งส่งตรวจจะถูกเพิ่มจำนวนโดยใช้ Primer ที่ติดฉลากเรืองแสงไว้ โดย Primer ที่ใช้จะมีหลายตัวซึ่งออกมาแบบมาจำเพาะต่อตำแหน่งบนโครโมโซมคู่ที่ 13, 18, และ 21 โดยเลือกใช้ประมาณ 3-4 ตำแหน่งบนโครโมโซม 1 แท่ง เพื่อช่วยในการยืนยันผลหากพบโครโมโซมแท่งนั้นเกิน จากนั้นชิ้นส่วน DNA ที่ได้จากการสังเคราะห์จะถูกนำมาวิเคราะห์โดย Capillary electrophoresis ซึ่งจะทำการคัดแยกชิ้นส่วนเหล่านี้ตามขนาด โดยชิ้นส่วนขนาดเล็กจะผ่านไปได้ก่อน และขนาดเท่ากันจะผ่านไปพร้อมๆกัน และสามารถบอกได้ว่าเป็นชิ้นส่วนมาจากโครโมโซมคู่ใดจากฉลากเรืองแสงที่ติดไว้ ดังนั้นจะสามารถบอกขนาดและปริมาณของชิ้นส่วนนั้นๆ จากนั้นข้อมูลเหล่านี้จะถูกนำไปวิเคราะห์เชิงปริมาณต่อ (Quantitative analysis) และแสดงผลออกมาในรูปแบบกราฟ (4)
การใช้ QF-PCR ในบทบาทการตรวจหา Aneuploidy จะให้ผลค่อนข้างเร็วภายใน 1-2 วัน เนื่องจากเซลล์ของทารกไม่จำเป็นต้องนำมาเพาะเลี้ยง อย่างไรการแปลผลนั้นจำเป็นต้องอาศัยผู้เชี่ยวชาญ และจำเป็นต้องมีการลงทุนทั้งทางด้านอุปกรณ์ และบุคลากรที่ผ่านการฝึกฝนมาแล้ว จากการศึกษาในประเทศสหรัฐอเมริกา พบว่าการตรวจนี้มีความน่าเชื่อถือร้อยละ 100 และไม่มีผลลบลวง (False negative) (5) จากการเปรียบเทียระหว่าง Karyotype กับ การทำ QF-PCR พบว่า ผลไปในทิศทางเดียวกัน ร้อยละ 98.75 และ พบว่า Karyotypeที่ผิดปกตินั้นไม่สามารถตรวจพบได้อีกร้อยละ 0.05 ดังนั้น QF-PCR จำสามารถให้ข้อมูลที่น่าเชื่อถือได้ทางคลินิก และลดขั้นตอนการทำงานของห้องปฏิบัติการ รวมไปถึงให้ผลที่เร็วกว่าสำหรับผู้ป่วยและแพทย์ผู้ดูแล (6)
ข้อเสียหลักๆของ QF-PCR คือ มีจุดอ่อนในการตรวจความผิดปกติของ Sex chromosome นั่นคือเมื่อ STRs ที่จำเพาะกับ Chromosome X ถูกใช้ QF-PCR อาจเเสดงผลออกมาเป็นลักษณะ Homozygous ของโครโมโซมเพศหญิงปกติ (XX) ซึ่งแยกไม่ได้กับลักษณะ Single X chromosome หรือ Monosomy X (XO) (5) อย่างไรก็ตามจุดอ่อนนี้สามารถแก้ไขโดยการใช้ Highly polymorphic marker ซึ่งมีความจำเพาะต่อ X แต่ละตัวแทนในการทดสอบ
การตรวจ Chromosomal microarray analysis หรือ CMA
การตรวจนี้มีความไวในการตรวจ (Sensitivity) มากกว่าการตรวจ Standard karyotyping ถึง 100 เท่า สามารถตรวจหา Microdeletion และ Microduplication ได้เล็กถึง 50-100 กิโลเบส การตรวจ Direct CMA ใช้เวลาออกผลประมาณ 3-5 วัน แต่ถ้าหากจำเป็นต้องเพาะเลี้ยงเซลล์เพิ่มอาจใช้เวลา 10-14 วัน(1) เทคนิคการตรวจ Microarray สามารถใช้ได้ทั้งวิธี Comparative genomic hybridization (CGH) platform และ วิธี Single nucleotide polymorphism (SNP) platform หรือใช้ทั้งสองวิธีร่วมกัน (2)
CGH microarray platform เป็นการเปรียบเทียบ DNA จากสิ่งส่งตรวจ กับ DNA ควบคุมที่เป็นปกติ โดยจะมีส่วนของชิป (CGH chip) ที่ประกอบไปด้วยชิ้นส่วนของลำดับ DNA ที่เป็นคู่สมกับ DNA ที่สนใจ (Oligonucleotide) โดยที่ดีเอ็นเอของทารก (Fetal DNA) จากน้ำคร่ำหรือชิ้นส่วนของเนื้อรก (Chorionic villi sampling) จะถูกติดฉลากโดยสีเรืองแสง (Fluorescent) และจับเข้ากับ DNA ที่เป็นคู่สมที่อยู่บนชิป ส่วน DNA ควบคุมที่เป็นปกติจะถูกติดฉลากสีเรืองแสงอีกสี และก็จะเข้าจับกับ DNA ที่เป็นคู่สมบนชิปเช่นเดียวกัน จากนั้นความเข้มของสีเรืองแสง (Intensity of fluorescent signal) ที่ติดฉลากกับ DNA ของสิ่งส่งตรวจ และ ที่ติดฉลาดกับ DNA ควบคุมจะถูกเปรียบเทียบกัน ซึ่งปกติควรจะมีความเข้มแสงเท่ากัน หากความเข้มแสงของสิ่งส่งตรวจมีมากกว่า นั่นหมายความว่าโครโมโซมจากสิ่งส่งตรวจน่าจะมีชิ้นส่วนของโครโมโซมที่สนใจเกิน หากน้อยกว่า หรือไม่มี นั้นหมายถึงการขาดหายไปของชิ้นส่วนโครโมโซมที่สนใจ(2)
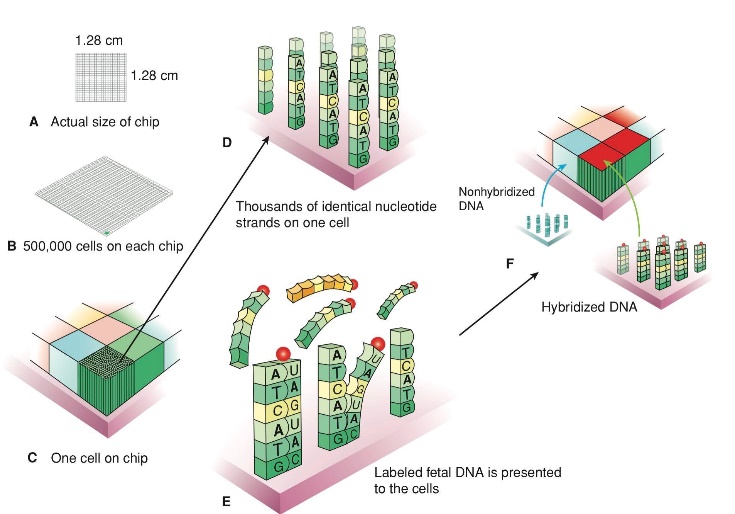 รูปที่ 3 ภาพแสดง Chromosomal microarray analysis
รูปที่ 3 ภาพแสดง Chromosomal microarray analysis
ภาพ A แสดงขนาดชิป
ภาพ B แสดงช่อง หรือที่เรียกว่าเซลล์ที่อยู่บนชิป
ภาพ C และ D แสดงภาพแต่ละช่อง ซึ่งภายในช่องจะมี Oligonucleotides นับพันที่เป็นคู่สมกับ DNA ที่สนใจที่หน้าตาเหมือนๆกันเคลือบอยู่บนผิว E แสดง DNA จากสิ่งส่งตรวจตัวอย่างที่ถูกติดฉลากและเข้าไปจับกับ DNA คู่สมบนชิป F เกิดการจับคู่กันจะเห็นสีที่ถูกติดฉลาก(2)
SNP array platform ในชิปจะประกอบไปด้วยลำดับ DNA ที่แตกต่างกัน โดยที่ปกติภายในสาย DNA ของสิ่งมีชีวิตสปีชีย์เดียวกันจะมีลำดับเบสบน DNA เหมือนกัน ยกเว้นเพียงตำแหน่ง SNP (Single nucleotide polymorphism)ซึ่งจะแตกต่างกันไปในแต่ละบุคคล เป็นตัวบอกอัตลักษณ์บุคคล โดยเมื่อ DNA ของทารกที่จับกับ DNA คู่สมบนชิป นิวคลีโอไทด์ที่ติดฉลากสีเรืองแสงจะเข้ามาจับกับตำแหน่ง DNA ทารกที่เป็นคู่สมกันตรงตำแหน่ง SNP ทำให้ทราบชนิดของ nucleotide ที่ตำแหน่ง SNP ของตัวอย่างสิ่งส่งตรวจ และความเข้มของสัญญาณสีเรืองแสงจะเป็นตัวบ่งบอกจำนวนชุดของความแตกต่าง (7, 8)
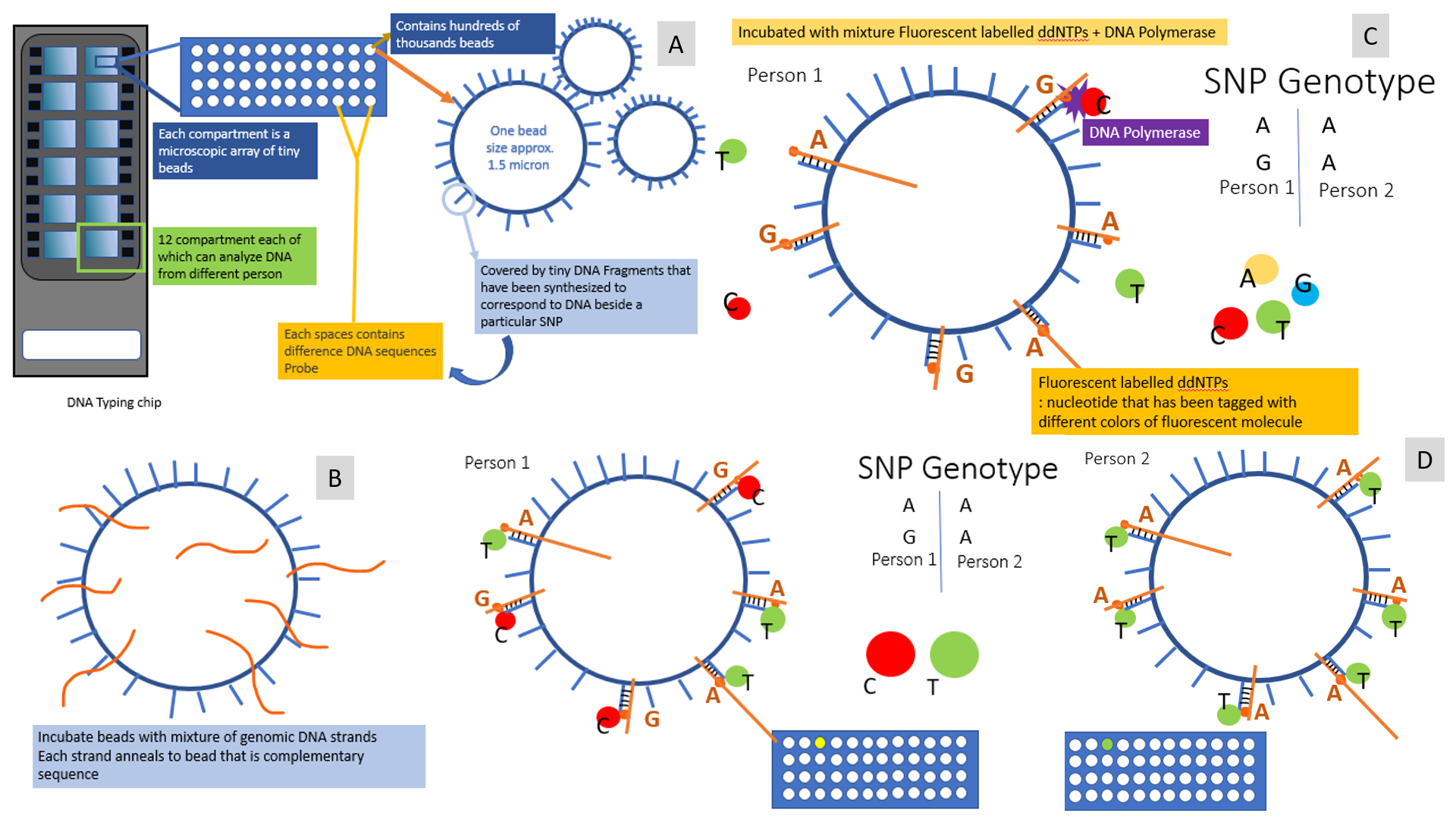
รูปที่ 4 แสดงชิปของ SNP microarray platform (ภาพ A) แสดงในหนึ่ง DNA typing chip จะประกอบไปด้วย 12 ส่วน เพื่อที่สามารตรวจได้ 12 ตัวอย่างพร้อมกัน ซึ่งแต่ละส่วนจะประกอบไปด้วยช่องเล็กๆ ที่ภายในหนึ่งช่องจะมีเม็ดบีด (Bead) หลายแสนตัว ซึ่งผิวของแต่ละเม็ดบีดจะห่อหุ้มด้วย DNA Probe ซึ่งจะมีลำดับ DNA ที่เป็นคู่สมกับ DNA ที่สนใจ โดยลำดับเบสที่อยู่บนผิวเม็ดบีดนั้นจะถึงแค่ตำแหน่งก่อนที่จะเป็นตำแหน่ง SNP โดยที่หากอยู่ในช่องเดียวกัน DNA probe ก็จะเป็นลำดับเบสเดียวกัน แต่ต่างกันไปในแต่ละช่อง จึงทำให้สามารถตรวจยีนได้หลายตำแหน่งในเวลาเดียวกัน (ภาพ B) ชิ้นส่วน DNA จากสิ่งส่งตรวจจะถูกผสมเข้าไปในช่องละจับกับ DNA probe ที่เป็นคู่สม (ภาพ C) ผสมนิวคลีโอไทด์ที่ติดฉลากสารเรืองแสงไว้เข้าไป โดย นิวคลีโอไทด์ที่เป็นคู่สมกับตำแหน่ง SNP จะเข้าไปจับกับตำแหน่ง SNP โดยมีเอนไซม์ DNA polymerase มาช่วยต่อเข้าไปในสาย (ภาพ D) จากบุคคลที่หนึ่งมี Genotype ที่ตำแหน่ง SNP เป็น heterozygous A/G ดังนั้น นิวคลีโอไทด์ที่เข้าไปจับจะมีสองส่วน และเห็นสีเรืองแสงสองสี ในขณะที่บุคคลที่สอง มีตำแหน่ง SNP เป็น homozygous A/A ดังนั้นก็จะแสดงสีเพียงแค่สีเขียว โดยที่ความเข้มของแสงจากแต่ละ bead จะแปลผลโดย high resolution scanning laser ดังนั้น เมื่อแปลผลทั้งชิป ซึ่งจะมีลำดับเบสที่แตกต่างกันไปในแต่ละช่อง จะสามารถรู้ลำดับเบสบน DNA ทั้งหมดของเกือบทุกโครโมโซมของสิ่งส่งตรวจ จึงสามารถบอกได้ว่ามีส่วนใดหายไป หรือส่วนใดเกิน โดยลำดับเบสที่จะทราบนั้นซึ่งขึ้นอยู่กับการออกแบบชิปของบริษัทที่ผลิตชิปว่าใส่ DNA probe ครอบคลุมลำดับเบสมากแค่ไหน ซึ่งในปัจจุบันลำดับเบสที่สามารถผลิตออกมาได้มีความครอบคลุมตำแหน่งที่สำคัญของทุกโครโมโซม (7)
ซึ่งทั้งสองเทคนิคสามารถตรวจหา Aneuploidy, Unbalanced translocations, Microdeletion และ Microduplication ได้ แต่ทั้งสองแบบจะไม่สามารถตรวจหา Balanced chromosomal rearrangement ได้ ด้วยเหตุผลนี้คู่แต่งงานที่มีปัญหาแท้งซ้ำซาก (Recurrent pregnancy loss) ควรได้รับการตรวจ Karyotype เป็นอันดับแรกก่อน (First line test) (2, 9)
นอกจากนี้ SNP array สามารถตรวจหาภาวะ Triploidy และ ภาวะ Absence of heterogenous ที่เกิดในกรณี Uniparental disomy ซึ่งคือการที่ทั้งสองแท่งของโครโมโซมคู่ใดคู่หนึ่งได้รับมาจากพ่อหรือแม่คนใดคนหนึ่งเท่านั้น เป็นภาวะที่มักขึ้นในภาวะ Trisomy rescue (2, 10)
การทำ Array อาจตรวจดูยีนโดยรวม (Genome-wide array) หรือการตรวจเพื่อพุ่งเป้าหมายไปที่กลุ่มโรคที่สงสัย (Targeted to known genetic syndrome) ก็ได้ ซึ่งการตรวจ Genome wide array มักใช้ในการศึกษาวิจัยมากกว่า ส่วนการตรวจ Targeted arrays จะมีบทบาทในการตรวจทารกในครรภ์ (Prenatal genetic test)(2, 11)
การประยุกต์ใช้ในทางคลินิก
ในหญิงตั้งครรภ์ที่มีความเสี่ยงสูงในการเกิด Autosomal trisomy จากการตรวจคัดกรองโครโมโซมที่ผิดปกติ (Aneuploidy screening) ควรได้รับการตรวจ Karyotyping หรือ การตรวจ FISH ร่วมกันกับ Karyotyping หากพบว่าทารกมีความผิดปกติ (Fetal abnormalities) แล้วทำการตรวจ Karyotyping ผลออกมาเป็นปกติ พบว่า Chromosomal microarray สามารถตรวจพบความผิดปกติได้ถึงร้อยละ 6.5 และ ร้อยละ 1-2 หากทารกไม่พบความผิดปกติที่ชัดเจน (12) ดังนั้น ACOG และ SMFM จึงแนะนำให้สามารถพิจารณาทำการตรวจ CMA เป็นการตรวจแรก (First tier test ) ถ้าหากพบว่าทารกมีความผิดปกติทางโครงสร้าง (fetal structural abnormalities) แทนการตรวจ Karyotyping แต่ถ้าหากความผิดปกติที่พบค่อนข้างจำเพาะกับกลุ่มโรค Aneuploidy ชัดเจน เช่น การพบ Endocardial defect ที่พบในกลุ่มโรค Trisomy 21 หรือการมี Alobar holoprosencephaly ที่พบใน Trisomy 13 การตรวจขั้นต้นก็ควรทำเป็น Karyotyping และ หรือร่วมกับ FISH (1, 13) โดยจำเป็นต้องให้ข้อมูลข้อดีและข้อจำกัดในการตรวจแต่ละชนิดเพื่อให้มารดาใช้ประกอบการตัดสินใจในการเลือก CMA อาจสามารถช่วยในการระบุโรคทางพันธุกรรมที่เป็นยีนส์เด่น (Autosomal dominant genetic disorders) แต่ยังไม่แสดงอาการในรุ่นพ่อแม่ที่มียีนส์ดังกล่าว และยังสามารถบ่งชี้ในกรณีที่โรคไม่ได้ถ่ายทอดจากรุ่นพ่อแม่สู่ลูก (2)
สำหรับการตรวจทารกที่มีภาวะตายคลอด (Stillbirth) การตรวจ CMA มักถูกใช้เป็นการตรวจมาตราฐานมากกว่าการตรวจ Standard karyotyping หนึ่งในเหตุผลคือการตรวจ CMA ไม่จำเป็นต้องใช้เซลล์ที่สามารถแบ่งตัวได้ จากข้อมูล The Stillbirth Collaborative Research Network พบว่าประมาณร้อยละ 6 ของเคสที่การตรวจ Karyotyping ไม่สามารถให้ข้อมูลได้ การตรวจ CMA สามารถระบุได้ว่าเป็น Aneuploidy หรือมี Pathologic copy number variant โดยที่โดยรวมแล้วการตรวจ CMA สามารถให้ผลได้มากกว่าการตรวจ Karyotyping เพียงอย่างเดียวถึงร้อยละ 25 (1, 13, 14)
การตรวจ CMA สามารถตรวจหาชิ้นส่วนของ DNA ที่มีการเพิ่มเติมหรือขาดหายที่มีขนาดเล็กได้ถึงอย่างน้อย 1000 คู่เบส ซึ่งชิ้นส่วนขนาดเล็กเหล่านี้ที่มีการเปลี่ยนแปลงเพิ่มเติม หรือขาดหายไปจากตัวอย่างปกติจะถูกเรียกว่า Copy number variants ซึ่งชิ้นส่วนนี้อาจมีนัยยะสำคัญทางคลินิกซึ่งสัมพันธ์กับภาวะปกติ (Benign copy number variants ) หรือ ภาวะผิดปกติ (Pathologic Copy number variants) ที่สัมพันธ์กับโรคทางพันธุกรรมซึ่งพบได้ถึงร้อยละ 15 (15) แต่ก็ยังมีส่วนของ Copy number variants เหล่านี้ที่ปัจจุบันยังไม่ทราบว่ามีนัยยะสำคัญทางคลินิกหรือไม่เนื่องจากยังไม่มีข้อมูลของตำแหน่งลำดับเบสเหล่านั้นว่าสัมพันธ์กับการแสดงออกทางพันธุกรรม (Phenotype) แบบใด ซึ่งจะถูกเรียกว่า Copy number variants of uncertain significance หรือ Variants of uncertain clinical significant (11)
การตรวจ CMA ยังสามารถระบุความผิดปกติของสารพันธุกรรมที่เป็นโรคที่เกิดในวัยผู้ใหญ่ (Adult onset disorder) เช่น โรค Charcot-Marie-Tooth disease ซึ่งเกิดจาก Duplication ที่อาจส่งต่อมาจากบิดา หรือ มารดาที่ไม่มีอาการ ดังนั้นจึงจำเป็นต้องอธิบายภาวะเหล่านี้ให้ผู้ป่วยรับทราบก่อนการส่งตรวจ (11)
Whole Genome sequencing และ whole exome sequencing [next generation sequencing]
โดยส่วนมากทารกในครรภ์ที่มีความผิดปกติทางโครงสร้าง (Structural abnormalities) มักมีผล Karyotype หรือ CMA ที่ปกติ การตรวจ Whole Genome sequencing เป็นเทคนิคการตรวจ genome ทั้งหมด ส่วน Whole exome sequencing เป็นการตรวจเฉพาะส่วนของ genome ที่จะแปลเป็น DNA ซึ่งคิดเป็นร้อยละ 1 ของ genome ทั้งหมด (2, 11) โดยจากการศึกษาพบว่าสามารถระบุความผิดปกติของสารพันธุกรรมในรายที่ทารกมีภาวะผิดปกติหลายส่วน (Multiple anomalies) ซึ่งไม่พบความผิดปกติจากการตรวจมาตรฐาน เช่น Karyotype , Microarray หรือทั้งสองอย่างได้ถึงร้อยละ 20-30 (16)
การใช้สองเทคนิคนี้มีการใช้มากขึ้นกับในการตรวจหลังคลอด เพื่อประเมินในกรณีสงสัยโรคทางพันธุกรรม(Genetic syndrome) หรือมีปัญหาด้านการเรียนรู้ (Intellectual disability) ในปัจจุบันมีการแนะนำให้ใช้ในกรณีที่ทารกในครรภ์สงสัยภาวะโรคทางพันธุกรรมชนิดใดชนิดหนึ่งเป็นพิเศษ ซึ่งไม่สามารถวินิจฉัยได้จากการตรวจ CMA ACOG แนะนำให้เลือกทำเป็นกรณีไป เช่น ในกรณีที่เด็กทารกมีลักษณะผิดปกติที่ไม่สามารถมีชีวิตรอดได้ หรือมีภาวะความผิดปกติที่ซ้ำกับทารกในครรภ์ก่อนหน้า (Lethal or recurrent anomalies) ที่การตรวจอื่นไม่สามารถให้ข้อมูลหรือวินิจฉัยได้(11) ข้อจำกัดของการตรวจเทคนิคนี้คือเวลาการตรวจที่อาจใช้เวลานาน ดังนั้น จึงยังไม่ได้มีการนำมาใช้ในส่วนของ Prenatal test มากนัก (2, 11)
อย่างไรก็ตามเนื่องจากผลการตรวจ CMA อาจพบผลที่เป็น Copy number variants of uncertain significance หรือ Variants of uncertain clinical significant ซึ่งจำเป็นต้องใช้ผู้เชี่ยวชาญในการแปลผล และผลนี้อาจทำให้เกิดความกังวลใจกับผู้ป่วยได้ ดังนั้นจำเป็นอย่างยิ่งที่จะต้องมีการอธิบายรายละเอียดรวมถึงผลที่อาจเกิดขึ้นได้กับผู้ป่วยก่อนทำการส่งตรวจ (11)
Fetal DNA in maternal circulation
เซลล์ของทารก (Fetal cells) จะอยู่ในเลือดมารดาในอัตราส่วนความเข้มข้นที่ค่อนข้างต่ำ ประมาณ 2-6 เซลล์/มิลลิลิตรของเลือดมารดา ซึ่งบางครั้งเซลล์เหล่านี้อาจอยู่ในเลือดแม่ค้างนานกว่าสิบปีหลังคลอด โดยที่เซลล์ที่ยังคงค้างอยู่นั้นอาจการแฝงเข้าไปในเซลล์มารดา (Engraft) และทำให้เกิดภาวะ Microchimerism ซี่งเชื่อว่าเป็นสาเหตุของโรคภูมิคุ้มกันต่อตนของมารดา (Maternal autoimmune disease) เช่น Scleroderma, SLE และHashimoto thyroiditis ในเวลาต่อมา สำหรับการตรวจเพื่อวินิจฉัยจากเซลล์ทารกในเลือดมารดานั้นถูกจำกัดโดยความเข้มข้นของปริมาณเซลล์ที่น้อย เซลล์จากการตั้งครรภ์ก่อนหน้านี้ และความยากในการแยกเซลล์ทารกกับเซลล์มารดา แต่ Cell free DNA นั้นสามารถ ก้าวข้ามผ่านข้อจำกัดเหล่านี้ได้(2)
Cell free DNA คือ ชิ้นส่วนของ DNA นี้ได้จากเซลล์ของมารดา (maternal cell) และ เซลล์รกที่มีการตาย (Apoptotic placental trophoblastic cell) ซึ่งมักถูกเรียกว่าเป็น DNA ของทารก โดย Cell free DNA จะมีความน่าเชื่อถือเมื่ออายุครรภ์มากกว่า 9-10 สัปดาห์ สัดส่วนของ cell free DNA ซึ่งมาจากรกนั้น ถูกเรียกว่า Fetal fraction โดยถือเป็นส่วนประกอบประมาณร้อยละ10 ของ Cell free DNA ทั้งหมดในพลาสมาของมารดา
Cell free DNA ต่างกันกับ Intact fetal cell ตรงที่สามารถล้างออกมาจากเลือดของมารดาโดยใช้เวลาภายในไม่กี่นาที สำหรับการศึกษาวิจัย Cell free DNA สามารถใช้เพื่อตรวจหา Single gene disorder ได้หลากหลายโรค ซึ่งส่งผ่านมาทาง Alleles ของพ่อ หรือ แม่ ได้แก่ Myotonic dystrophy, Achondroplasia, Huntington disease, Congenital adrenal hyperplasia, Cystic fibrosis และ alpha thalassemia(9) การนำไปใช้ในทางคลินิกของ cell-free DNA ได้แก่ การตรวจคัดกรอง Aneuploidy, การบอกเพศทารก และการบอกหมู่เลือด Rh (Rh D genotyping) (2)
การคัดกรอง Aneuploidy
มี 2-3 วิธีที่แตกต่างในการตรวจคัดกรองเด็กทารก Trisomy และ Sex chromosomal aneuploidies โดยใช้ cell free fetal DNA ได้แก่ วิธีการ Sanger sequencing, Whole genome sequencing ที่มักถูกเรียกว่า Massive parallel หรือ Short gun sequencing วิธีการเลือกดูเฉพาะโครโมโซม (Chromosome selective) หรือ Targeted sequencing และ วิธี SNPs analysis (Allele analysis)โดยสามารถรู้สัดส่วนของชิ้นส่วนโครโมโซมแต่ละคู่ว่าสูงเกินที่ควรจะเป็นหรือไม่ ดังนั้น มารดาที่มีทารกในครรภ์เป็นโรคดาว์ซินโดรมจะมีสัดส่วนของ DNA จากโครโมโซมคู่ที่ 21 มากกว่าปกติ
การตรวจคัดกรองจาก Cell free DNA นั้นจาก 37 การศึกษาพบว่ามีความไว (Sensitivity) ในการตรวจพบ Down syndrome อยู่ที่ร้อยละ 99 และการตรวจหา trisomy 18 และ 13 อยู่ที่ ร้อยละ 96 และ 91 และ ความจำเพาะ (Specificity) อยู่ที่ ร้อยละ 99.9 ผลบวกลวง (False positive) ของการตรวจคัดกรอง Aneuploidy อยู่ที่น้อยกว่าร้อยละ 1 โดยในปัจจุบัน การตรวจ Cell free DNA เพื่อคัดกรอง ถูกแนะนำให้เป็นทางเลือกในมารดาที่มีความเสี่ยงมากขึ้นที่ทารกจะเป็น Fetal autosomal trisomy (2, 9)
ในบางครั้ง cell free DNA screen อาจไม่สามารถบอกผลได้ ในเคสร้อยละ 4-8 ซึ่งอาจจะเกิดจากความล้มเหลวของ assay, high assay variance หรือ low fetal fraction
การบอกเพศของเด็กทารก
ในมุมมองโรคทางพันธุกรรม การบอกเพศของเด็กทารกนั้นอาจมีประโยชน์หากทารกนั้นมีความเสี่ยงในการเกิดโรค X-linked disorder และ ยังมีประโยชน์หากทารกมีความเสี่ยงในการเป็น Congenital adrenal hyperplasia เนื่องจาก การให้ Corticosteroid ในแม่อาจหลีกเลี่ยงที่จะให้หากทารกนั้นเป็นเพศชาย ในการศึกษาในหญิงตั้งครรภ์มากกว่า 6,000 คน ความไว (Sensitivity) ในการบอกเพศจาก Cell free fetal DNA อยู่ที่ ร้อยละ 95 โดยตรวจช่วงอายุครรภ์ 7-12 สัปดาห์ และมากถึงร้อยละ 99 หากตรวจหลัง 20 สัปดาห์ ดังนั้นจึงถือเป็นทางเลือกหนึ่งที่ใช้แทนการตรวจรุกล้ำ (Invasive testing ) (2)
การบอกหมู่เลือด Rh D Genotype
ในกลุ่มประชากรผิวขาวเกือบร้อยละ 40 ของเด็กทารกที่มารดาเป็น Rh D negative จะมีหมู่เลือดเป็น Rh D negative เช่นเดียวกัน การตรวจโดยใช้ cell free DNA สามารถทำได้โดยการใช้ real time PCR ต่อ exons ของ
RHD gene ซึ่งมักจะอยู่ที่ exon 4, 5 และ 7 ซึ่งการตรวจพบว่าทารกนั้นเป็น Rh D negative เช่นเดียวกันตั้งแต่ต้นจะช่วยหลีกเลี่ยงการตรวจ Middle cerebral artery doppler และการเจาะน้ำคร่ำที่ไม่จำเป็น และช่วยลดค่าใช่จ่ายรวมถึงความเสี่ยงจากการทำหัตถการ (2)
การตรวจหมู่เลือด Rh ด้วยวิธีการ Cell free DNA จะถูกทำเป็นปกติในประเทศเดนมาร์คและเนเธอร์แลนด์ ซึ่งจะคัดกรองในหญิงตั้งครรภ์ที่อายุครรภ์ 27 สัปดาห์ พบว่าผลลบลวง (False negative) อยู่ที่ร้อยละ 0.03 และ ผลบวกลวง (False positive) อยู่ที่น้อยกว่าร้อยละ 1 (2)
ตารางสรุปเปรียบเทียบเทคนิคการตรวจทางพันธุศาสตร์ของทารกในครรภ์แต่ละชนิด(1)
| การทดสอบ | เวลาที่ใช้ | ภาวะที่ตรวจพบได้ | หมายเหตุ |
| Conventional karyotype (Cultured cell – metaphase analysis) | 7-14 วัน | โครโมโซมที่ผิดปกติที่ขนาดมากว่า 5-10 เมกะเบส | |
| FISH
-Direct preparation (Interphase) ไม่ต้องเพาะเลี้ยงเซลล์ |
24-48 ชั่วโมง | การตรวจที่เร็วสำหรับ ภาวะ Aneuploidy หลัก ๆ เช่น โครโมโซม 13, 18, 21 , X และ Y | การตรวจ FISH จากโดยวิธีไม่เพาะเลี้ยงเซลล์จากการเจาะชิ้นเนื้อรกมีความแม่นยำน้อยกว่าจากการเพาะเลี้ยงเซลล์ที่ได้จากชิ้นเนื้อรก หรือน้ำคร่ำ ดังนั้นหากพบความผิดปกติควรได้รับการยืนยันจาการทำ FISH จากเซลล์ที่เพาะเลี้ยงซ้ำ |
| FISH
-Cultured cells (Metaphase) |
7-14 วัน | Microdeletion และ duplication | ใช้ในภาวะผิดปกติที่จำเพาะ (specific abnormalities) เมื่อมีข้อมูลทางคลินิกที่สงสัย |
| QF-PCR | 1-2 วัน | การตรวจที่เร็วสำหรับ ภาวะ Aneuploidy หลัก ๆ เช่น โครโมโซม 13, 18, 21 , X และ Y | |
| Chromosomal microarrays | 3-5 วัน (ไม่เพาะเลี้ยงเซลล์)
10-14 วัน (เพาะเลี้ยงเซลล์) |
ชิ้นส่วนของDNA ที่เพิ่มขึ้น หรือ หายไป (copy number of variants) ที่มีขนาดมากกว่า 50-200 กิโลเบส | เป็นการตรวจทั้งจีโนม เพื่อหา copy of variants โดยที่จะไม่สามารถตรวจหา Balanced rearrangement และ Triploidies การตรวจจะมีความหลากหลายเนื่องจาก microarray platform ที่ออกแบบมาต่างกัน |
| Molecular DNA testing | 3-14 วัน เร็วกว่าหากไม่ได้มีการเพาะเลี้ยงเซลล์ | ตรวจหา Genetic mutation ที่รู้ล่วงหน้าอยู่แล้ว หรือเคยมีการแสดงออกมาแล้วในครอบครัว หรือสงสัยจากการตรวจภาพอัลตราซาวด์ หรือจากสิ่งตรวจพบอื่นของทารกในครรภ์ | Targeted test focusing on specific disorder ซึ่งส่วนใหญ่จะได้หลักฐานข้อมูลจากประวัติครอบครัว หรือลักษณะของทารกจาก อัลตราซาวด์ |
ในปัจจุบันเทคนิคการตรวจเพื่อคัดกรองทางพันธุศาสตร์ซึ่งเป็นที่นิยมใช้คือ Cell free fetal DNA เนื่องจากมีความไวและความจำเพาะในการตรวจค่อนข้างสูงรวมถึงหลีกเลี่ยงการทำหัตการลุกล้ำโดยไม่จำเป็น อย่างไรก็ตามหากพบความผิดปกติจากการส่งตรวจข้างต้นก็จำเป็นที่จะต้องส่งตรวจทางพันธุศาสตร์เพิ่มเติมเพื่อการวินิจฉัย โดย ACOG แนะนำให้ทำการตรวจ CMA เป็นการตรวจแรกถ้าหากพบว่าทารกมีความผิดปกติทางโครงสร้างแทนการตรวจ Karyotyping เนื่องจากสามารถตรวจพบความผิดปกติได้ถึงร้อยละ 6.5 ในกรณีที่ทารกมีความผิดปกติทางโครงสร้างและตรวจ Karyotype ให้ผลปกติ แต่ถ้าหากความผิดปกติที่พบค่อนข้างจำเพาะกับกลุ่มโรค Aneuploidy ชัดเจนการตรวจขั้นต้นก็ควรทำเป็น Karyotyping และ หรือร่วมกับ FISH แต่ไม่แนะนำให้ทำการวินิจฉัยจากผลการตรวจ FISH เพียงอย่างเดียว การตัดสินใจควรมีองค์ประกอบของผลตรวจ FISH ร่วมกับการตรวจอย่างน้อยข้อหนึ่งดังต่อไปนี้ การตรวจวินิจฉัยทางพันธุกรรม Karyotyping หรือ การตรวจ Chromosomal microarray หรือ ข้อมูลทางคลินิกที่ช่วยส่งเสริม เช่น การตรวจคัดกรองภาวะ Aneuploidy ที่ผิดปกติ หรือ ภาพถ่ายอัลตราซาวด์ เทคนิค QF-PCR ก็ถือเป็นอีกวิธีที่สามารถให้ผลที่มีความน่าเชื่อถือในกรณีสงสัยภาวะ Aneuploidy ของโครโมโซม 13,18,21 ซึ่งสามารถให้ผลตรวจที่เร็วภายใน 1-2 วัน แต่ก็จำเป็นต้องอาศัยผู้เชี่ยวชาญในการแปลผล ส่วน Whole Genome sequencing และ whole exome sequencing [Next generation sequencing] ACOG แนะนำให้เลือกทำเป็นกรณีไป เช่น ในกรณีที่เด็กทารกมีลักษณะผิดปกติที่ไม่สามารถมีชีวิตรอดได้ หรือมีภาวะความผิดปกติที่ซ้ำกับทารกในครรภ์ก่อนหน้า (Lethal or recurrent anomalies) ที่การตรวจมาตรฐานอื่น เช่น Karyotype , CMA หรือทั้งสองอย่างไม่สามารถให้ข้อมูลหรือวินิจฉัยได้ และก่อนการส่งตรวจไม่ว่าจะเป็นการส่งตรวจใดทุกครั้งควรมีการให้คำอธิบายถึงวิธี ผลการตรวจที่อาจเกิดขึ้นได้ ข้อดีข้อเสีย และข้อจำกัดของแต่ละวิธีให้กับผู้ป่วยทุกครั้ง
เอกสารอ้างอิง
1. American College of Obstetricians, Gynecologists. Practice bulletin no. 162: Prenatal diagnostic testing for genetic disorders. Obstetrics and gynecology. 2016;127(5):e108-e22.
2. Cunningham FG, Leveno KJ, bloom SL, Dashe JS, Hoffman BL, Casey BM, et al. Williams Obstetrics. 25 ed: McGraw-Hill Education; 2018.
3. Schrijver I, Zehnder JL. Tools for genetics and genomics: Cytogenetics and molecular genetics.
4. Hixson L, Goel S, Schuber P, Faltas V, Lee J, Narayakkadan A, et al. An overview on prenatal screening for chromosomal aberrations. Journal of Laboratory Automation. 2015;20(5):562-73.
5. Brown L, Abigania M, Warburton D, Brown S. Validation of QF‐PCR for prenatal aneuploidy screening in the United States. Prenatal Diagnosis: 2006;26(11):1068-74.
6. Badenas C, Rodríguez-Revenga L, Morales C, Mediano C, Plaja A, Pérez-Iribarne MM, et al. Assessment of QF-PCR as the first approach in prenatal diagnosis. The Journal of Molecular Diagnostics. 2010;12(6):828-34.
7. Kim S, Misra A. SNP Genotyping: Technologies and Biomedical Applications. Annu Rev Biomed Eng. 2007;9:289-320.
8. He C, Holme J, Anthony J. SNP genotyping: the KASP Assay. Crop Breeding: Springer; 2014. p. 75-86.
9. Rose NC, Kaimal AJ, Dugoff L, Norton ME, American College of Obstetricians, Gynecologists. Screening for fetal chromosomal abnormalities: ACOG practice bulletin, number 226. Obstetrics & Gynecology. 2020;136(4):e48-e69.
10. Chau MHK, Choy KW. The role of chromosomal microarray and exome sequencing in prenatal diagnosis. Current Opinion in Obstetrics and Gynecology. 2021;33(2):148-55.
11. Obstetricians ACo, Gynecologists. Microarrays and next-generation sequencing technology: the use of advanced genetic diagnostic tools in obstetrics and gynecology. ACOG Committee opinion no. 682. American College of Obstetricians and Gynecologists. Obstet Gynecol. 2016;128:e262-e8.
12. Callaway JL, Shaffer LG, Chitty LS, Rosenfeld JA, Crolla JA. The clinical utility of microarray technologies applied to prenatal cytogenetics in the presence of a normal conventional karyotype: a review of the literature. Prenatal Diagnosis. 2013;33(12):1119-23.
13. Hay SB, Sahoo T, Travis MK, Hovanes K, Dzidic N, Doherty C, et al. ACOG and SMFM guidelines for prenatal diagnosis: Is karyotyping really sufficient? Prenatal diagnosis. 2018;38(3):184-9.
14. Levy B, Wapner R. Prenatal diagnosis by chromosomal microarray analysis. Fertility and Sterility. 2018;109(2):201-12.
15. Vissers LE, Veltman JA, van Kessel AG, Brunner HG. Identification of disease genes by whole genome CGH arrays. Human Molecular Genetics. 2005;14(suppl_2):R215-R23.
16. Drury S, Williams H, Trump N, Boustred C, GOSGene, Lench N, et al. Exome sequencing for prenatal diagnosis of fetuses with sonographic abnormalities. Prenatal Diagnosis. 2015;35(10):1010-7.


